Formeln des Lebens
Auch in biologischen Systemen gilt die Physik. KI besitzt dieses Vorwissen jedoch meist nicht. Deshalb besteht die Gefahr, dass sie bei der Beschreibung von Organismen zu unsinnigen Ergebnissen kommt. Der Informatiker Ivo Sbalzarini und seine Forschungsgruppe entwickeln KI, die physikalische Gesetzmäßigkeiten in die Analyse biologischer Abläufe einbezieht und so Biologie und Mechanik miteinander verbindet. Auf diese Weise lässt sich herausfinden, wie Gene die Gestalt eines Gewebes beeinflussen. Auch könnten sich damit künftig Medikamente gezielter entwickeln lassen.
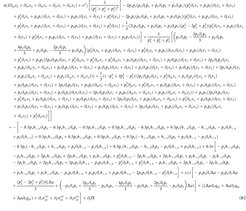
Auf den Punkt gebracht
- KI mit physikalischem Vorwissen: Algorithmen, die biologische Prozesse beschreiben sollen, müssen die physikalischen Gesetzmäßigkeiten berücksichtigen, die vor Ort herrschen.
- Neue Darstellung von Bildern: Um die Arbeit der Algorithmen effizienter zu machen, haben die Forschenden eine inhaltsadaptive Bilddarstellung namens Adaptive Particle Representation (APR) entwickelt.
- Zusammenführung von Daten: Die Synthese unterschiedlicher biologischer Daten mithilfe von KI kann fundamental neue Erkenntnisse zum Zusammenhang zwischen Genen und Funktion eines Organismus liefern und die Entwicklung von Medikamenten erleichtern.
Text: Nora Lessing
Die Frage nach dem Leben, dem Universum und dem ganzen Rest? „42“, lautet die Antwort eines Hochleistungsrechners in Per Anhalter durch die Galaxis. Im Roman des britischen Autors Douglas Adams führt das verblüffende Ergebnis zum Bau des Supercomputers Erde. Das Ziel: Den Wortlaut der ursprünglichen Frage zu rekonstruieren.
Die Biologie als gigantisches, sich selbst steuerndes Rechensystem – da würde Ivo Sbalzarini sofort mitgehen. Der Informatiker leitet die universitäre Abteilung am Zentrum für Systembiologie Dresden, eine Forschungsgruppe am Max-Planck-Institut für molekulare Zellbiologie und Genetik, und hält eine Informatikprofessur an der Technischen Universität Dresden. Nicht einverstanden wäre er dagegen mit unklaren Eingaben und unverständlichen Ergebnissen wie in Adams Roman. Im Gegensatz zu dessen Protagonisten wissen der Forscher und sein Team ganz genau, wie die von ihnen entwickelten Algorithmen zu ihren Ergebnissen kommen, denn diese basieren auf nachvollziehbaren mathematischen Konzepten und physikalischen Prinzipien wie zum Beispiel der Massenerhaltung oder dem Kräftegleichgewicht. „Da unsere KI nur innerhalb dieser Leitplanken aktiv ist, kann sie zwar nicht so kreativ sein wie zum Beispiel Sprachmodelle wie ChatGPT, dafür sind ihre Ergebnisse leichter interpretierbar, und es kommt nicht zu den gefürchteten Halluzinationen. Das macht sie für das wissenschaftliche Verständnis zu einem wertvollen Werkzeug“, erklärt Ivo Sbalzarini.
Das Leben ist berechenbar
Ein heranwachsender Fischembryo; eine Pflanze, die ihre Blüte öffnet; weiße Blutkörperchen, die Viren den Garaus machen – in jedem Organismus laufen Abertausende unterschiedlicher Vorgänge ab. So kompliziert das Geschehen auch sein mag, es ist trotzdem berechenbar. Bis vor wenigen Jahren waren unsere Möglichkeiten dazu jedoch stark begrenzt. Zwar wurden seit den 1940er-Jahren Algorithmen entwickelt, die Vorgänge in der Kernphysik und Strömungsmechanik berechnen. Diese sollten die Physik der Atomspaltung simulieren oder die Flugbahn von Artilleriegeschossen bestimmen.
Lebende Systeme jedoch bedürfen andersartiger physikalischer Beschreibungen. Ein Beispiel dafür ist die in der Beschreibung toter Materie oft gemachte Gleichgewichtsannahme, der zufolge sich tote Materie im thermodynamischen Gleichgewicht befindet oder dieses anstrebt. Deshalb kühlt ein Becher Kaffee ab, wenn man ihn stehen lässt, ein Stück Metall beginnt nicht spontan zu hüpfen. Lebewesen dagegen können eine konstante Körpertemperatur beibehalten und sich aus eigenem Antrieb bewegen. Sie können das tun, weil sie über ihre Nahrung Energie zugeführt bekommen. Die Beschreibung solcher Nicht-Gleichgewichtssysteme unterscheidet sich fundamental von der klassischer physikalischer Gleichgewichtsgesetze. Entsprechend ungeeignet sind herkömmliche Algorithmen für deren Berechnung.
Materialwissenschaft des Lebens
Um der Komplexität des Lebens eine mathematisch berechenbare Ordnung abzutrotzen, müssen KI-Algorithmen her. „Wir wollen die Prinzipien der Selbstorganisation lebender Materie entschlüsseln – man könnte das auch als Materialwissenschaft des Lebens bezeichnen“, sagt Sbalzarini. Dabei gehen die Forschenden ganzheitlich vor. Sie betrachten also nicht isolierte Einzelkomponenten wie Proteine, Gene oder Wachstumsfaktoren, sondern konzentrieren sich in ihrer Arbeit darauf, wie diese innerhalb biologischer Strukturen wie Zellen und Geweben miteinander interagieren. „Dabei ist uns die Mechanik sehr wichtig – zum Beispiel, wie weich oder wie flüssig die Materialien sind und welchen physikalischen Prinzipien sie gehorchen müssen.“
Dazu braucht es einen mathematischen Blick auf die Welt. Menschen erfahren die Umwelt durch ihre Sinne: Wenn sie trockene Erde zwischen ihren Fingern zerreiben etwa, wenn sie in eine saftige Aprikose beißen oder über den Anblick bizarr geformter Pilze staunen. Sbalzarini und sein Team betrachten das Leben aber noch aus einer anderen Perspektive: Sie gehen davon aus, dass die Natur eine durchweg berechenbare Angelegenheit ist. Hinter der Vielfalt der Natur entdecken sie mathematische Gleichungen, die komplexe biologische Vorgänge modellieren. Die Forschenden aus Dresden überführen das sinnlich Erfahrbare also in eine Welt aus Zahlen. Dabei helfen ihnen ihre neu entwickelten KI-Algorithmen.
Vorhersage biologischer Abläufe
Am Beispiel eines sich entwickelnden Fadenwurmembryos lässt sich das Prinzip veranschaulichen, nach dem Ivo Sbalzarini und sein Team vorgehen. Biologinnen und Biologen studieren mittels sogenannter Lichtblattmikroskopie, wie der Embryo im Laufe seiner Entwicklung wächst und neue Strukturen bildet. Aus den Daten findet eine von der Arbeitsgruppe Sbalzarini entwickelte KI die Gleichungen, die diese Entwicklung und die dabei wirkenden Kräfte mathematisch beschreiben. Damit können sie von jedem Zeitpunkt aus vorhersagen, wie die weitere Entwicklung ablaufen wird. Auch die Aktivität von Genen oder die Bewegung von Zellen im Embryo könnte die KI beschreiben. „Wir suchen also die Gesetzmäßigkeit, der die Entwicklung folgt. Damit können wir den Vorgang beschreiben und vorhersagen, wie er weiter ablaufen wird“, erklärt Sbalzarini. Anhand experimenteller Daten aus den Laboren ihrer Projektpartner können er und sein Team überprüfen, ob der Algorithmus mit seiner Prognose richtig liegt. Die neuen KI-Algorithmen haben hier die Erfolgsquote enorm gesteigert.
Für viele biologische Prozesse ist es bislang jedoch noch nicht gelungen, entsprechende Gleichungen zu finden. Für manche aber durchaus. Allerdings sind die von der KI gefundenen Gleichungen manchmal derart kompliziert, dass sie nur schwer gelöst werden können. Aber auch hier helfen neue Algorithmen „made in Dresden“. So kann das Team von Ivo Sbalzarini jetzt zum Beispiel die Gleichungen lösen, die das Wachstum und die selbstständige Bewegung lebender Gewebe beschreiben.
Lebende Gewebe verhalten sich wie sogenannte aktive polare Gele. Das sind Materialien, die sich aus eigenem Antrieb („aktiv“) bewegen können, die aber weder flüssig noch fest sind. Ein nicht-aktives Beispiel ist Zauberknete: Zu einem Ball geformt und auf den Boden geworfen, springt sie wie ein Flummi weg. Einfach nur auf den Tisch gelegt, zerfließt sie langsam. „Lebendes Gewebe verhält sich genau so: Wird es rasch verformt, nimmt es wieder seine ursprüngliche Form an. Wenn es dagegen langsam deformiert wird, passt es sich an. Und aktiv ist es auch noch“, erklärt Sbalzarini.
Lösungen für Gleichungen

Die physikalischen Prinzipien, die die Mechanik aktiver polarer Gele beschreiben, sind seit über 20 Jahren bekannt. Aber lösen konnte man die daraus resultierenden komplizierten mathematischen Gleichungen bislang nur in vereinfachten Spezialfällen. Nach jahrelanger Forschung hat die Arbeitsgruppe um Ivo Sbalzarini kürzlich einen Algorithmus vorgestellt, der diese Gleichungen lösen kann. Damit lassen sich die dreidimensionale Form lebender Materialien und die Kräfteverteilungen, die in ihnen herrschen, sehr genau berechnen. Nun wollen die Forschenden den Algorithmus erweitern, um auch Vorgänge in beweglichen oder wachsenden biologischen Strukturen vorhersagen zu können.
Wenn die KI aus den Bildern eines Lichtblattmikroskops physikalische Modelle ableitet, muss sie allerdings riesige Datenmengen verarbeiten. Die sind mit gängigen Methoden oft nicht zu bewältigen. Die Arbeitsgruppe von Ivo Sbalzarini hat deshalb auch einen Algorithmus entwickelt, mit dem sich Bilder effizienter speichern und analysieren lassen. Bis vor Kurzem war es eine technische Herausforderung, dreidimensionale Aufnahmen von lebenden biologischen Geweben zu analysieren.
In der Lichtblattmikroskopie werden die Fluoreszenzmoleküle in einzelnen hauchdünnen Schichten des zu untersuchenden Objekts nacheinander von einem Laser zum Leuchten angeregt. Die Bilder aus den verschiedenen Schichten werden anschließend zu einem 3D-Bild zusammengefügt. Die so entstehenden Aufnahmen sind besonders detailreich, aber sie erzeugen Tera- oder sogar Petabytes an Daten. „Das Hochladen der Bilder auf einen Server wird damit zur Geduldsprobe. Das teure Mikroskop kann in der Zeit nicht anderweitig genutzt werden. Und wenn die Daten auf dem Server angekommen sind, kann man die Bilder noch nicht mal anschauen, weil sie so groß sind, dass sie in keinen Grafikkartenspeicher passen. Das ist wie ein Ferrari, der nur auf Feldwegen fahren kann und dann auch noch alle paar Meter im Matsch stecken bleibt.“
Relevante und irrelevante Pixel
Deshalb haben Ivo Sbalzarini und sein Team eine Technik entwickelt, die große Bilder sehr viel effizienter verarbeiten und damit für KI nutzbar machen kann. Mit dieser APR – Adaptive Particle Representation – genannten Technik erstellte Aufnahmen besitzen dieselbe Qualität wie herkömmliche Pixel-Bilder, sie benötigen jedoch nur einen Bruchteil des Speicherplatzes und der Rechenzeit. Anders als bei herkömmlichen Bildern wird im neuen Verfahren Information nur dort gespeichert, wo sie auch vorhanden ist. In einem Bild eines Fruchtfliegenembryos vor einem schwarzen Hintergrund zum Beispiel benötigt jeder Pixel gleich viel Speicherplatz – und das obwohl der Embryo und weniger der Hintergrund das Interessante ist. „Meistens sind nicht alle Pixel in einem Bild relevant für dessen Informationsinhalt“, erklärt Sbalzarini. Die APR findet die informativen Stellen in einem Bild und speichert Helligkeit oder Farbe nur dort.
Anders als bei Komprimierungsprogrammen, mit denen sich Bilder auch platzsparend speichern lassen, müssen APR-Bilder vor der weiteren Bearbeitung nicht erst wieder dekomprimiert – also entpackt – werden. Dadurch spart man nicht nur Festplattenspeicher, sondern auch Rechenzeit. Außerdem wird der Arbeitsspeicher der Grafikkarte entlastet, sodass die Bilder schneller angeschaut und analysiert werden können, als das Mikroskop sie aufnimmt. „Mit anderen Worten: Wir haben dem Ferrari eine Autobahn gebaut, damit er nun Maximalgeschwindigkeit fahren kann. Und die inhaltsadaptive APR-Darstellung hilft auch der KI, denn sie muss nicht erst lernen, wo die relevante Information sich im Bild versteckt.“
Kommerzielle Nutzung von APR

Die APR-basierte Mikroskopie hat Sbalzarini mithilfe von Max-Planck-Innovation, der Verwertungsgesellschaft der Max-Planck-Gesellschaft, zum Patent angemeldet. Zwei ehemalige Mitglieder der Arbeitsgruppe haben zudem ein Start-up mit Sitz im englischen Oxford gegründet, das die APR für alle Formen der Fluoreszenzmikroskopie kommerziell verfügbar machen soll. Aber auch für andere Bildgebungsverfahren, wie etwa für die Radioastronomie, könnte die Technik eines Tages interessant sein. Bereits heute verwenden Biologinnen und Biologen außerhalb Dresdens die APR, um die Bilder ihrer Hochleistungsmikroskope zu analysieren. Das verbessert die Auslastung der Mikroskope, spart Speicherplatz, Zeit und Strom. Zugleich können sie damit dreidimensionale Bilder in Echtzeit in Virtual Reality darstellen. Forschende in Genf haben auf diese Weise zum Beispiel für eine fluoreszenzmikroskopische 3D-Aufnahme der Nervenzellen in einem kompletten Mausgehirn lediglich eine Stunde und zwanzig Minuten Rechenzeit benötigt. Ohne die neue Technik hätte das vier Tage gedauert.
Auch dank der APR-Bilddarstellung ist die KI bei der physikalischen Beschreibung des Lebens schon ein gutes Stück vorangekommen. Ivo Sbalzarini ist überzeugt: „Mit physikalischem Vorwissen ausgestattete KI wird in den nächsten Jahren entscheidende Fortschritte in der Systembiologie möglich machen. Denn wenn ein neuronales Netz ausschließlich Modelle lernen kann, die die im jeweiligen Fall gültigen physikalischen Prinzipien nicht verletzen, lassen sich viel unnötige Rechnerei und realitätsfremde Ergebnisse vermeiden.“ Zugleich würden die KI-Modelle dadurch erklärbarer, zuverlässiger und ressourcensparender.
Kleine Datensätze, riesige Datenpunkte
Die Entwicklung von KI zur Analyse biologischer Abläufe bleibt aber eine Mammutaufgabe. Teil des Problems sind die Daten selbst. „Für die Entwicklung von Sprachmodellen gibt es riesige Datensätze. Die einzelnen Datenpunkte – zum Beispiel Wortgruppen – sind aber klein. In der Systembiologie ist es oft umgekehrt: Die Datensätze sind klein, einige wenige Mikroskopbilder von der Entwicklung eines Embryos. Die einzelnen Datenpunkte – ein einzelnes Bild – sind dagegen riesig. Das macht das Training von KI-Systemen für die räumliche Biologie so schwierig.“
In den kommenden Jahren wollen Ivo Sbalzarini und sein Team KI, Physik und Biologie noch näher zusammenbringen. Passend dazu hat die Max-Planck-Gesellschaft in Kooperation mit der Boehringer Ingelheim Stiftung und der Technischen Universität Dresden eine zusätzliche Abteilung für biomedizinische KI am Zentrum für Systembiologie Dresden geschaffen. 20 Millionen Euro ist das der Boehringer Ingelheim Stiftung wert. Weitere 20 Millionen steuern die Max-Planck-Gesellschaft, die Technische Universität Dresden und der Freistaat Sachsen bei. Nach 10 Jahren übernimmt die Max-Planck-Gesellschaft die Kosten.
Das wissenschaftliche Konzept für die neue Abteilung stammt maßgeblich aus Sbalzarinis Feder. Es geht darum, unter Einbezug physikalischer Prinzipien Datensätze zusammenzuführen und für KI nutzbar zu machen. „Genetik, Biochemie, Zellbiologie und Entwicklungsbiologie, um nur eine kleine Auswahl zu nennen, haben enormes Wissen angehäuft. Wir kennen zum Beispiel das gesamte Proteininventar eines Organismus, die Aktivität seiner Gene, die Formveränderungen während der Entwicklung und so weiter. Aber niemandem ist es bislang gelungen, all diese Datensätze miteinander zu verbinden und so das Gesamtsystem zu verstehen. Weil die Datensätze so groß, heterogen und komplex sind, schaffen wir das nur mithilfe von KI.“
Vom Genotyp und Phänotyp
Mikroskopische Aufnahmen, Proteinkonzentrationen, Gen-Daten – die Dresdner Forschenden wollen für all das eine gemeinsame Sprache finden und es mittels KI zusammenführen. Das klingt so, als könnte man auf diese Weise das Leben in die digitale Welt überführen und gewissermaßen „digitale Zwillinge“ biologischer Abläufe oder sogar Organismen erzeugen. Aber das ist nicht das eigentliche Ziel. „Die Synthese unterschiedlichster Datentypen könnte uns helfen zu verstehen, wie die Gene und die Funktion eines Organismus miteinander zusammenhängen. Dieser Zusammenhang zwischen Genotyp und Phänotyp ist bis heute unklar“, sagt Sbalzarini. Auch für die Pharmaforschung könnte das ein großer Fortschritt sein: Wenn die Abläufe in biologischen Systemen dank KI vorhersagbar werden, könnten pharmazeutische Wirkstoffe gezielt und effizienter entwickelt werden.
Bereits heute steht außer Frage, dass viele Algorithmen dazu gar nicht erst neu erfunden werden müssen: Das Leben selbst hat sie vor langer Zeit hervorgebracht – in Form biochemischer Netzwerke in Zellen. Wie Algorithmen werten diese Informationen aus ihrer Umwelt aus, lernen dazu und reagieren immer wieder unterschiedlich auf Veränderungen. „Anders als in digitalen Computern gibt es dabei aber nicht nur die Spannungszustände 0 und 1, sondern unermesslich viele Kombinationen aus Konzentrationen Zehntausender Proteine. In gewisser Weise funktioniert das Leben auf der Erde also wie ein gigantischer Quantencomputer“, resümiert Ivo Sbalzarini. Ein Vergleich ganz im Sinne von Douglas Adams und seinem Roman.














